Segundo o psicólogo e filósofo Carl Gustav Jung, a dúvida é o cerne do crescimento pessoal.
As questões metabólicas – sobre o metabolismo dos seres vivos – são praticamente infinitas, posto que a maioria dos organismos ainda é desconhecida. Estima-se que menos de 3% de todas espécies de bactérias e 5% dos fungos da Terra são conhecidos.
Dificuldades relacionadas à descrição da biodiversidade das bactérias marinhas constituem enormes desafios a serem superados. Por exemplo, utilizando-se análises de sequências de DNA ribossômico 16S depositadas no GenBank, observou-se que espécies marinhas de bacterioplâncton correspondem a 1.117 ribotipos únicos, dos quais 609 foram atribuídos a clones ambientais não cultivados e a 508 de bactérias cultivadas (Hagström et al., 2002). Gêneros e espécies completamente novas de Actinomycetaceae marinhos foram descobertos, tal como o táxon MAR 1, de ampla ocorrência em regiões tropicais e subtropicais. O táxon MAR 1, pertencente ao clado Micromonosporaceae, é constituído por estirpes que nem sempre exigem condições estritamente salinas para crescimento. Estas linhagens foram repetidamente isoladas em coletas seqüenciais de sedimentos marinhos. Linhagens de MAR 1 são dominantes, correspondendo a ca. 90% dos Actinomycetae encontrados em sedimentos marinhos. Os novos clados MAR 2 e MAR 3, relacionados a Streptomyces spp., também foram isolados (Jensen et al., 2005). Muitas das novas cepas de MAR 2 e MAR 3 foram atribuídas a novos gêneros, como Salinispora (Maldonado et al., 2005) e Marinispora (Kwon et al., 2006; Ward e Bora, 2006).
Espécies descritas de fungos marinhos atualmente correspondem a 537; porém, o número real deve ultrapassar 10.000 espécies. É ainda necessário se investigar fungos marinhos de todos os tipos de substratos, com o objetivo de obter uma estimativa real de sua diversidade. Estes substratos incluem manguezais, de algas marinhas, animais marinhos, sedimentos e espécies encontradas em alto mar e mar profundo (Gareth Jones, 2011).
Cepas de fungos do solo parecem ser muito mais abundantes do que em qualquer outro ambiente (Schmidt et al., 2007; Schmidt et al., 2008). Considerando que aproximadamente 80.000-100.000 espécies descritas de fungos provavelmente correspondem a apenas 5% de todas as espécies (Schmidt et al., 2008), é evidente que existe uma diversidade inimaginável de cepas de fungos a serem descobertas, descritas e bioprospectadas.
Bactérias do solo também são abundantes, correspondendo a 10e9 células bacterianas por grama de solo. No entanto, estas são difíceis de cultivar em meios artificiais (Janssen, 2008). É consenso que 95% dos fungos ainda não foram cultivados, ou seja, são essencialmente desconhecidos (Demain e Sanchez, 2009; Hawksworth, 2001; Kis-Papo, 2005). Estimativas de 1,5 milhões de espécies de fungos foram propostas em 1991 (Hawksworth, 1991), atualizadas para 2,3 milhões de espécies em 2001 (Hawksworth, 2001) e revisadas até 3,0 milhões de espécies em 2012 (Hawksworth, 2012), um nível impressionante de diversidade. Outros autores sugerem 611.000 espécies de fungos na Terra, sendo 7% conhecidas (Mora et al., 2011), 712.000 de espécies de fungos totais (Schmit e Muller, 2007). No entanto, esse número de espécies parece ser subestimado (Hawksworth, 2012). Os dados da diversidade de fungos indicam que esta é maior nas regiões temperadas, embora a dimensão dessa diferença seja difícil de estabelecer. Hawksworth (2012) afirma que “a falta de estimativas abrangentes da relação fungo:planta nos trópicos continua a ser um problema, e esses estudos são vitais (…)”. Dados de sequenciamento de genomas de alto rendimento (high throughtput genome sequencing) indicam uma diversidade de fungos ainda mais impressionante, com correlação direta entre diversidade de espécies e precipitação de chuva no solo, mas não necessariamente entre espécies de fungos e diversidade de espécies de plantas (McGuire et al., 2011). Estudos realizados na floresta amazônica colombiana durante 3,5 anos levou ao isolamento de 632 espécies de fungos macroscópicos, dos quais 52% não puderam ser descritos em nível de espécie (López-Quintero et al., 2012). Outros grupos de fungos, mais específicos, são abundantes e em grande parte desconhecidos em regiões tropicais (Hawksworth, 2012). Quando ferramentas moleculares foram empregadas para investigar relação numérica fungo:planta, o número parece ser próximo de 8 para avaliações realizadas no Reino Unido e no Alasca (Howksworth, 2012). Howksworth sugere uma análise cuidadosa de fatores ambientais que podem determinar a extensão e a natureza da comunidade de fungos, e que esses números de diversidade fúngica devem ser cuidadosamente considerados dentro de um intervalo entre 1,5 e 3 milhões de espécies (Howksworth, 2012).
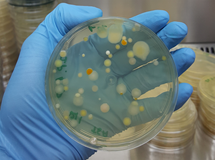
A diversidade das bactérias do solo também é excepcionalmente alta. Avaliação pioneira da diversidade bacteriana do solo por análise do DNA do solo obtida em Seim, Norte de Bergen, Noruega, apresentou uma diversidade de cerca de 4.000 genomas bacterianos distintos, correspondendo a uma diversidade aproximadamente 200 vezes maior do que de bactérias cultiváveis (Torsvik et al., 1990). Análises de impressão digital do DNA ribossômico da diversidade bacteriana de 96 amostras de solo coletadas na América do Norte e do Sul demonstrou que o pH do solo é o fator mais importante que influencia a diversidade bacteriana. Não foi observada correlação entre a diversidade de bactérias e plantas, ou com temperatura, gradiente latitudinal ou distância geográfica (Fierer e Jackson, 2006). Avaliação recente, utilizando-se metagenômica, demonstrou que a diversidade bacteriana do solo varia tanto vertical quanto horizontalmente, e sua avaliação é metodologicamente muito dependente. Os resultados mostram que procedimentos metagenômicos apenas fornecem resultados tendenciosos da comunidade bacteriana do solo, e que esta pode ser muito maior do que o número atualmente aceito, entre 10e4 a 10e7 espécies por grama de solo (Delmont et al., 2011) .
Apesar da diversidade de micro-organismos de solo e sua versatilidade metabólica, nos últimos anos essa diversidade microbiana tem sido negligenciada. Muita ênfase tem sido direcionada para a descoberta de novas linhagens microbianas e produtos naturais bioativos de endófitos, micro-organismos marinhos e extremófilos; a bio- e quimiodiversidade dos micro-organismos do solo foram praticamente “esquecidas” após muitos anos de prospecção bem-sucedida, particularmente de antibióticos, mas também de outros agentes quimioterapêuticos (Clardy et al., 2006). Muitos pesquisadores acreditam que a prospecção de micro-organismos de solo está “esgotada”, uma vez que o grau de redundância na descoberta de produtos naturais bioativos já conhecidos das cepas microbianas do solo é extremamente elevado. Todavia, o que é evidente é que os bioprospectores microbianos do solo é que estão esgotados, uma vez que são necessárias inovações metodológicas, esforços contínuos e dedicação para se desenvolver novos procedimentos e novas abordagens utilizando-se tecnologia de ponta, de maneira a melhor se compreender e explorar os micro-organismos terrestres como fonte de compostos bioativos (Pearce et al., 2010). Micro-organismos de solo, particularmente actinomicetos, são excelentes produtores de produtos químicos bioativos estruturalmente únicos. Estes incluem antibióticos bacterianos e fúngicos, agentes anticancerígenos, inibidores enzimáticos, imunossupressores, drogas hipocolesterolêmicas, inseticidas e agentes antiparasitários (Demain e Sanchez, 2009; Clardy et al., 2006; Singh et al., 2010 Genilloud et al., 2011).
Assim, é mais do que evidente que a maior parte do metabolismo secundário ainda é completamente desconhecida.
Referências
Clardy, J., Fischbach, M.A., Walsh, C.T. (2006) New antibiotics from bacterial natural products. Nature Biotechnol., 24:1541-155.
Delmont, T.O., Robe, P., Cecillon, S., Clark, I.M., Constancias, F., Simonet, P., Hirsch, P.R., Vogel, T.M. (2011) Accessing the Soil Metagenome for Studies of Microbial Diversity. Appl. Environm. Microbiol., 77:1315-1324.
Demain, A.L., Sanchez, S. (2009) Microbial drug discovery: 80 years of progress. J. Antibiot., 62:5-16.
Fierer, N., Jackson, R. B. (2006) The diversity and biogeography of soil bacterial communities. Proc. Natl. Acad. Sci. USA., 103 626-631.
Gareth Jones, E.B. (2011) Are there more marine fungi to be described? Bot. Mar., 54:343–354.
Genilloud, O., González, I., Salazar, O., Martín, J., Tormo, J. R., Vicente, F. (2011) Current approaches to exploit actinomycetes as a source of novel natural products. J. Ind. Microbiol. Biotechnol., 38, 375-389.
Hagström, A., Pommier, T., Rohwer, F., Simu, K., Stolte, W., Svensson, D., Zweifel, U.L. (2002) Use of 16S Ribosomal DNA for Delineation of Marine Bacterioplankton Species. Appl. Environm. Microbiol., 68:3628-3633.
Hawksworth, D.L. (1991) The fungal dimension of biodiversity: magnitude, significance, and conservation. Mycol. Res., 95:641–655.
Hawksworth, D.L. (2001) The magnitude of fungal diversity: the 1.5 million species estimate revisited. Mycol. Res., 105:1422–1432.
Hawksworth, D.L. (2012) Global species numbers of fungi: are tropical studies and molecular approaches contributing to a more robust estimate? Biodiv. Conserv., 21, 2425–2433.
Janssen, P.H. (2008) New cultivation strategies for terrestrial microorganisms, in Accessing Uncultivated Microorganisms: from the Environment to Organisms and Genomes and Back. Ed. by K. Zengler. ACM Press, Washington, DC, 2008, 173-192.
Kis-Papo, T. (2005) Marine Fungal Communities, in The Fungal Community – Its Organization and Role in the Ecosystem. Dighton, J., White, J.F., Oudemans, P. (eds.). CRC Press, Taylor & Francis, Boca Raton, Florida, 80.
Kwon, H.C., Kauffman, C.A., Jensen, P.R., Fenical, W. (2006) Marinomycins A−D, Antitumor-Antibiotics of a New Structure Class from a Marine Actinomycete of the Recently Discovered Genus “Marinispora”. J. Am. Chem. Soc., 128:1622–1632.
López-Quintero, C.A., Straatsma, G., Franco-Molano, A.E., Boekhout, T. (2012) Macrofungal diversity in Colombian Amazon forests varies with regions and regimes of disturbance. Biodiv. Conserv., 21, 2221-2243.
McGuire, K.L., Fierer, N., Bateman, C., Treseder, K.K., Turner, B.L. (2011) Fungal Community Composition in Neotropical Rain Forests: the Influence of Tree Diversity and Precipitation. Microb. Ecol., 63:804-812.
Pearce, C., Eckard, P., Gruen-Wollny, I., Hansske, F.G. (2010) Microorganisms: their role in the discovery and development of medicines, in Natural Product Chemistry for Drug Discovery. Buss, A.D. and Butler, M.S. (eds.). RSC Publishing, Cambridge, UK, 215-241.
Schmidt, S. K., E. K. Costello, D. R. Nemergut, C. C. Cleveland, S. C. Reed, M. N. Weintraub, A. F. Meyer, and A. M. Martin. (2007) Biogeochemical consequences of rapid microbial turnover and seasonal succession in soil. Ecology, 88, 1379-1385.
Schmidt, S.K., Wilson, K.L., Meyer, A.F., Schadt, C.W., Porter, T.M., Monclavo, J.M. (2008) The Missing Fungi: New Insights from Culture-Independent Molecular Studies of Soil, in Acessing Uncultivated Microorganisms: from the Environment to Organisms and Genomes and Back. Ed. by Zengler,K. ACM Press, Washington, DC, 55-66.
Singh, B.K., Macdonald, C.A. (2010) Drug discovery from uncultivable microorganisms. Drug Discov. Today, 15:792-799.
Torsvik, V., Goksøyr, J., Daae, F. L. (1990) High diversity in DNA of soil bacteria. Appl. Environ. Microbiol., 56, 782-787.
Ward, A.C., Bora, N. (2006) Diversity and biogeography of marine actinobacteria. Curr. Opin. Microbiol., 9, 279–286.
Curtir isso:
Curtir Carregando...
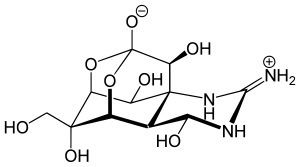
 Mais fascinante do que o isolamento de um composto tão polar sem a ajuda de técnicas cromatográficas foi sua determinação estrutural, realizada sem a utilização de ressonância magnética nuclear (RMN) ou de espectrometria de massas (EM). A estrutura da TTX foi elucidada principalmente por métodos de degradação química e análises por ultravioleta e infravermelho, tendo sido confirmada por análise por difração de raios X. O mais interessante é o fato de que 4 grupos de pesquisa apresentaram, de maneira simultânea e independente, a estrutura da TTX no mesmo simpósio de produtos naturais da IUPAC, realizado em 1964 em Kyoto, no Japão: os grupos de Bob Woodward (Pure Appl. Chem., 1964, 9, 49; J. Am. Chem. Soc., 1964, 86, 5030); K. Tsuda (Chem. Pharm. Bull., 1964, 12, 642; idem, ibidem, 1964, 12, 1257), Mosher (Science, 1964, 144, 1100) e Goto (Tetrahedron, 1965, 21, 2059). Subsequentemente, mais de 20 derivados da TTX foram isolados das mais diversas fontes: salamandras, sapos, polvos, caranguejos, algas, além de muitos outros, sendo que mais recentemente se descobriu que a verdadeira origem da TTX é bacteriana. Muitas revisões foram escritas sobre a TTX. Uma particularmente interessante foi publicada por John Daly no Journal of Natural Products (2004, 67, 1211-1215). Ainda não se conhece a origem biossintética da TTX.
Mais fascinante do que o isolamento de um composto tão polar sem a ajuda de técnicas cromatográficas foi sua determinação estrutural, realizada sem a utilização de ressonância magnética nuclear (RMN) ou de espectrometria de massas (EM). A estrutura da TTX foi elucidada principalmente por métodos de degradação química e análises por ultravioleta e infravermelho, tendo sido confirmada por análise por difração de raios X. O mais interessante é o fato de que 4 grupos de pesquisa apresentaram, de maneira simultânea e independente, a estrutura da TTX no mesmo simpósio de produtos naturais da IUPAC, realizado em 1964 em Kyoto, no Japão: os grupos de Bob Woodward (Pure Appl. Chem., 1964, 9, 49; J. Am. Chem. Soc., 1964, 86, 5030); K. Tsuda (Chem. Pharm. Bull., 1964, 12, 642; idem, ibidem, 1964, 12, 1257), Mosher (Science, 1964, 144, 1100) e Goto (Tetrahedron, 1965, 21, 2059). Subsequentemente, mais de 20 derivados da TTX foram isolados das mais diversas fontes: salamandras, sapos, polvos, caranguejos, algas, além de muitos outros, sendo que mais recentemente se descobriu que a verdadeira origem da TTX é bacteriana. Muitas revisões foram escritas sobre a TTX. Uma particularmente interessante foi publicada por John Daly no Journal of Natural Products (2004, 67, 1211-1215). Ainda não se conhece a origem biossintética da TTX.


























Você precisa fazer login para comentar.